技术摘要:
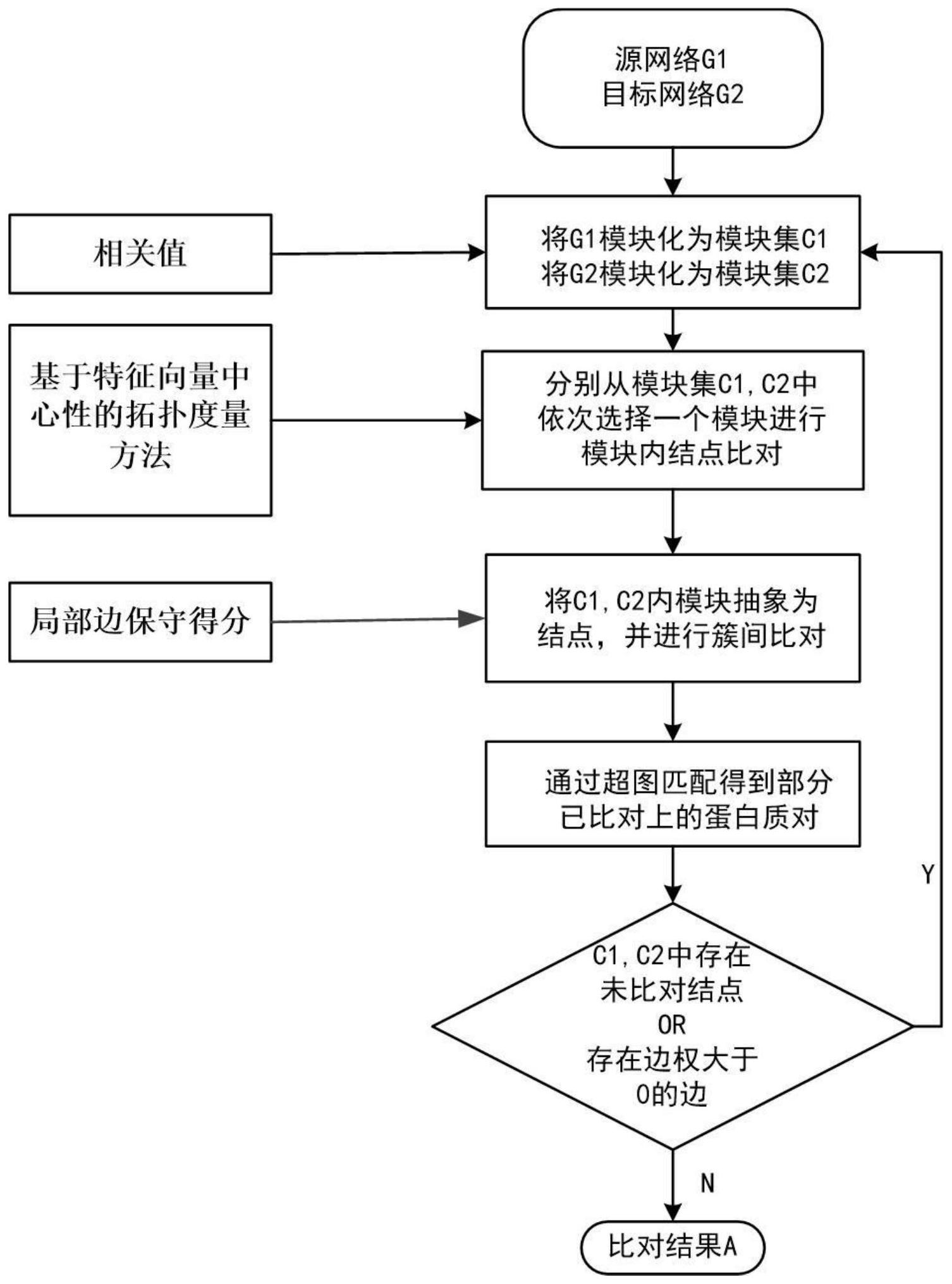
本发明公开了一种融合多种拓扑信息的生物网络比对方法。本发明一种融合多种拓扑信息的生物网络比对方法,包括:步骤1读取网络及其序列相似性得分,分别计算两个网络的相关值矩阵,并对网络进行模块划分,同一模块内的结点具有较高的相似性;步骤2计算模块内结点的相似 全部
背景技术:
近年来,随着生物实验的发展,产生了大量的生物网络数据,使用网络比对对生物 网络进行分析,比较不同物种间的相互作用,可以更好地理解物种间的进化关系,发现保守 的功能成分和实现功能预测。本发明研究两个网络的比对,两个网络分别为源网络和目标 网络。源网络结点数目比目标网络少,且其结点功能均为已知的;目标网络中蛋白质的功能 均未知。其生物意义为通过成对网络比对实现源网络与目标网络之间的功能转移,从而更 好的挖掘目标网络中蛋白质的功能。网络比对算法由得分函数和搜索算法两部分组成,得 分函数用于评价结点之间的相似性;搜索算法则根据得分函数搜索全局相似性最大的比对 结果。 目前已有的搜索算法主要分为二步算法和基于目标函数的搜索算法,二步算法的 第一步是计算两个不同输入网络的结点相似性,从而得到一个结点相似性得分矩阵。根据 这个相似性得分矩阵将网络比对问题转化为最大权重的二部图匹配问题,其中第一步计算 的结点相似性得分就是二部图的权重。二步算法的第二步,通过以前的经典算法或是贪婪 算法来求解最大权重的二部图匹配问题。第二步中抽取比对结果的策略决定了最终比对的 质量。大多数被提出的经典算法都属于二步算法。例如,IsoRank、MI-GRAAL、SPINAL以及 HubAlign。基于目标函数的搜索算法首先提出一个目标函数,然后再利用各种不同的启发 式搜索策略去优化目标函数。属于这类的算法有SANA,MAGNA,MAGNA 等。得分函数分为拓 扑得分和生物得分,目前已有的拓扑得分计算方法有度,GDV,Importance等,序列得分一般 采用由BLAST 产生的序列相似性得分。 传统技术存在以下技术问题: 1、现有技术对结点拓扑信息的挖掘不充分,导致最终比对的生物功能质量很高, 但拓扑质量很差。 2、现有技术通过模块信息来挖掘节点间的生物相似性,但模块化方法的选取不恰 当,导致产生错误的生物相似性得分,从而使其生物功能质量降低。 3、现有技术模块化时需要使用的blast序列相似性得分获取困难,大部分的数据 库仅包含不同网络中结点间的blast序列相似性得分,很少包含同一网络结点间的blast序 列相似性得分。 4、现有技术都存在拓扑与生物质量不平衡问题,一种质量的提高往往意味着另一 种质量的下降。
技术实现要素:
本发明要解决的技术问题是提供一种融合多种拓扑信息的生物网络比对方法, 5 CN 111599405 A 说 明 书 2/9 页 (1)充分挖掘结点间的拓扑相似性,以提高比对的拓扑质量。(2)模块化方法的确定,以保证 比对的生物功能质量不降低。(3)普适性的提高,如何解决同一网络中结点间的blast序列 相似性得分缺少问题。(4)拓扑与生物质量不平衡问题的解决,以便使在一种质量指标不降 低的前提下提高另一种质量指标。 为了解决上述技术问题,本发明提供了一种融合多种拓扑信息的生物网络比对方 法,包括: 步骤1读取网络及其序列相似性得分,分别计算两个网络的相关值矩阵,并对网络 进行模块划分,同一模块内的结点具有较高的相似性; 步骤2计算模块内结点的相似性得分,并对模块进行两两结点比对; 步骤3计算模块间的相似性得分,并对模块进行比对; 步骤4将步骤2,3中得到的结点映射关系进行整合,筛选,得到1对1的结点映射关 系; 步骤5删除已比对结点,并重复步骤2-5,小网络中的结点全部被比对上,或模块间 相似性得分为0,算法停止。 在其中一个实施例中,模块划分具体如下: 模块划分是针对单个网络进行的; 首先,计算网络的相关值矩阵,该矩阵给出了结点间的相似性关系,本发明给出了 结点间关系的四种定义,分别为强相关,弱相关,相关与不相关; 如果两个结点间有一条边相连,则称该对结点为强相关; 若结点间不存在直接相连的边,但可以通过其他结点间接相连,则称为弱相关; 符合强相关和弱相关的结点也称为相关; 不存在相关关系的结点对均称为不相关; 相关值计算公式如下: 其中,Θ强相关结点集,Φ为弱相关结点集, 为不相关结点集;max{1,|Φ*|M}指 所有相关结点中所经过的中间边数目的最大值,|Φ*(u,v)|指从结点u到v所经过的中间边数 目;公式(1)为归一化后的相关值,其值越大,表示结点间的相似性越高; 然后,根据相关值矩阵,分别对G1,G2进行模块化分,得到模块集合CG1,CG2;详细步 骤如下: a)构建关于网络G=(V,E)中所有结点对的相关值矩阵Ψ; b)对于 初始化|V|个分别以 为模块中心的 模块,记为 c)模块 的构建方法为:根据Ψ,得到其他结点与 的相关值,并将其按降序 排列,选取相关值在前25%的结点加入到模块 其他模块构造方法类似,最终得到 6 CN 111599405 A 说 明 书 3/9 页 在其中一个实施例中,模块内结点比对具体如下: 将网络G1,G2分别模块化后得到两个模块集合C1,C2;将C1中的每个模块与C2中的 每个模块分别使用种子扩展方法进行比对,得到|C1|*|C2|对模块比对结果,|C1|,|C2|分 别指模块的数目; 其中模块比对过程中用到的结点相似性得分函数为: 为结点(s,t)间的总相似性得分,B(s,t)为结点(s,t)的序列相似性得分,该 得分由BLAST 工具计算得出,用以评价结点间的生物相似性,值越大,结点相似性越高; 为结点间的拓扑相似性得分,它由一种基于特征向量中心性的拓扑向量元组 计算而来,其中 1) 表示结点 的度,即 的邻居数; 2) 表示结点 的特征向量中心性,用以衡量结点在网络中的中心性地位; 3) 表示结点 邻居的平均特征向量中心性; 因此,结点对(s,t)的拓扑相似得分 具体计算方式公式如公式(3);其值越 小,结点间越相似; 使用种子扩展方法将CG1中的模块分别与CG2中的模块两两进行模块内比对的详细 步骤如下: a)输入待比对模块 b)首先将 比对上; c)分别获取 的邻居, d)计算 中结点对的相似性 并使用匈牙 利算法将 结点进行比对,其中 为 与 的笛卡尔乘积; e)将已扩展结点 移除,并对剩余已比对结点对依次重复步骤c)d); f)获得模块内结点比对结果 在其中一个实施例中,模块间比对具体如下: 将每个模块看作一个结点,构建完全二部图,边的权重为模块间的相似性得分;接 着使用最大加权二部图匹配算法进行模块匹配,得到模块间比对结果;其中模块间相似性 得分计算如下: 为步骤2中得到的一个模块内比对结果中所比对上的结点对数目, 为该比对结果中结点对的序列相似性之和。 7 CN 111599405 A 说 明 书 4/9 页 在其中一个实施例中,其中, 为模块间的局部边保守得 分,用以衡量该比对结果的边保守性,具体计算如下: 令eij表示网络Gi中模块C(j)的局部边集,Ei为Gi的边集,V(C(j))为模块C(j)的结 点集,eij表示如下: ei,j={(s1,s2)|s1,s2∈V(C(j))∧(s1,s2)∈Ei} 5) 对于网络G1=(V1,E1),G2=(V2,E2), 如 果 则称 为一对模 块保守边; 表示模块 的保守边逻辑矩阵,其每个元素计算方式如下: 模块 的局部边保守得分计算如下: 在其中一个实施例中,其中,根据模块相似性得分对CG1,CG2进行模块间比对的详 细步骤如下: a)输入网络G1,G2的模块集合CG1,CG2; b)将CG1,CG2的每一个模块分别看作一个结点,构建完全二分图 边的权重为相似 性得分 c)使用匈牙利算法对 进行求解,即可得到一对一的模块比对 在其中一个实施例中,已有比对数据处理具体如下: 将已有的结点映射关系构建超图,超图的结点为已比对的结点,每对模块的比对 结果抽象为超图的一条超弧,使用超图匹配算法得到1对1的结点映射关系。 基于同样的发明构思,本申请还提供一种计算机设备,包括存储器、处理器及存储 在存储器上并可在处理器上运行的计算机程序,所述处理器执行所述程序时实现任一项所 述方法的步骤。 基于同样的发明构思,本申请还提供一种计算机可读存储介质,其上存储有计算 机程序,该程序被处理器执行时实现任一项所述方法的步骤。 基于同样的发明构思,本申请还提供一种处理器,所述处理器用于运行程序,其 中,所述程序运行时执行任一项所述的方法。 本发明的有益效果: (1)从网络结构、度、结点邻居、特征向量中心性、局部边保守等方面充分挖掘了结 点的拓扑相似性,提高了比对的拓扑质量。 (2)选取了合适的模块化方法,并提出了相关值概念,保证了比对的生物功能质 量。 (3)提出了相关值概念,解决了同一网络中结点间的blast序列相似性得分缺少问 8 CN 111599405 A 说 明 书 5/9 页 题,提高了算法的普适性。 (4)三种相似性得分函数的提出,保证了在提高拓扑质量的同时,算法的生物功能 质量不降低。 附图说明 图1是本发明融合多种拓扑信息的生物网络比对方法的流程图。 图2是本发明融合多种拓扑信息的生物网络比对方法的结点间相似性关系示例 图。 图3是本发明融合多种拓扑信息的生物网络比对方法的本发明与AligNet的比较 结果示意图。 图4是本发明融合多种拓扑信息的生物网络比对方法的本发明与其他算法的拓扑 质量图。 图5是本发明融合多种拓扑信息的生物网络比对方法的本发明与其他算法的生物 功能质量图。 图6是本发明融合多种拓扑信息的生物网络比对方法的本发明与其他算法的综合 表现。