技术摘要:
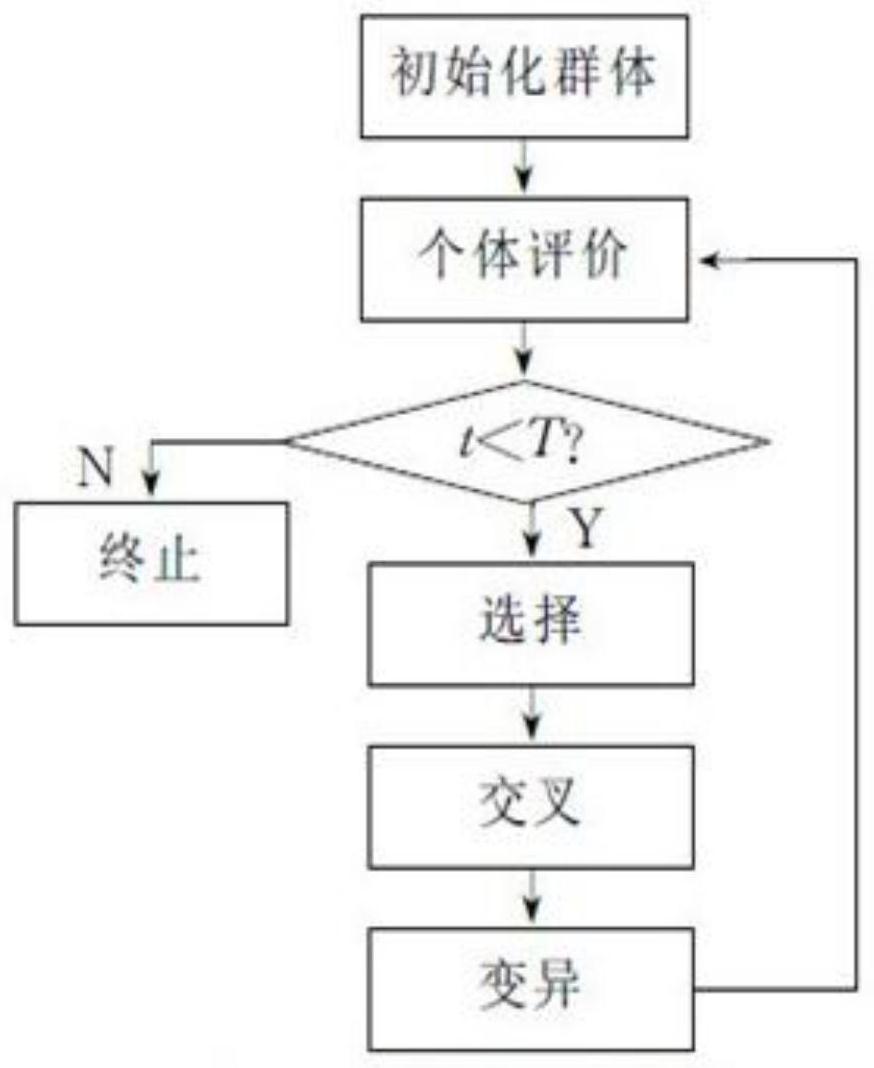
本发明公开一种基于Radi‑sharding架构数据副本的方法,包括如下步骤:(1)初始化种群;(2)交叉;(3)变异;(4)选择。本发明的算法给出了已经实现的复制方案以及特定对象的读写请求的变化,它可以快速调整副本分布以反映新的需求,AGRA运行的搜索空间缩小,使其能够在动 全部
背景技术:
在大型分布式系统中快速传播和访问信息已成为常见的应用场景。然而,终端依 然经常面临着在网络高峰时段的长时间延迟问题。在多个站点放置数据副本是减少网络流 量的一种有效的解决方案。跨读取密集型网络的数据副本可以潜在地增加流量,从而间接 减少终端用户的响应时间。近些年来,互联网在高峰时段经历的高下载时间激发了人们对 通过网络副本数据对象的兴趣,并提供了允许用户请求重定向到地理上接近的副本的机 制。 当前的趋势是以服务器镜像的形式执行粗粒度副本,即副本Web服务器的所有内 容。随着网络变得越来越大并且缓存限制变得明显,有理由相信细粒度副本(例如副本页 面)的重要性将增加。由于需要更新副本,维护大量副本可能会有性能瓶颈。传统分布式系 统中的副本,有三个主要问题,即分配,一致性和容错。虽然在web环境下,一致性和容错性 不是最重要的,但数据分配仍然是一个挑战。传统的简单副本算法(SRA)和基于遗传的启发 式算法(GRA)都是静态算法。一旦获得对象的副本分发,访问模式的改变将导致算法从头开 始运行,非常耗时。
技术实现要素:
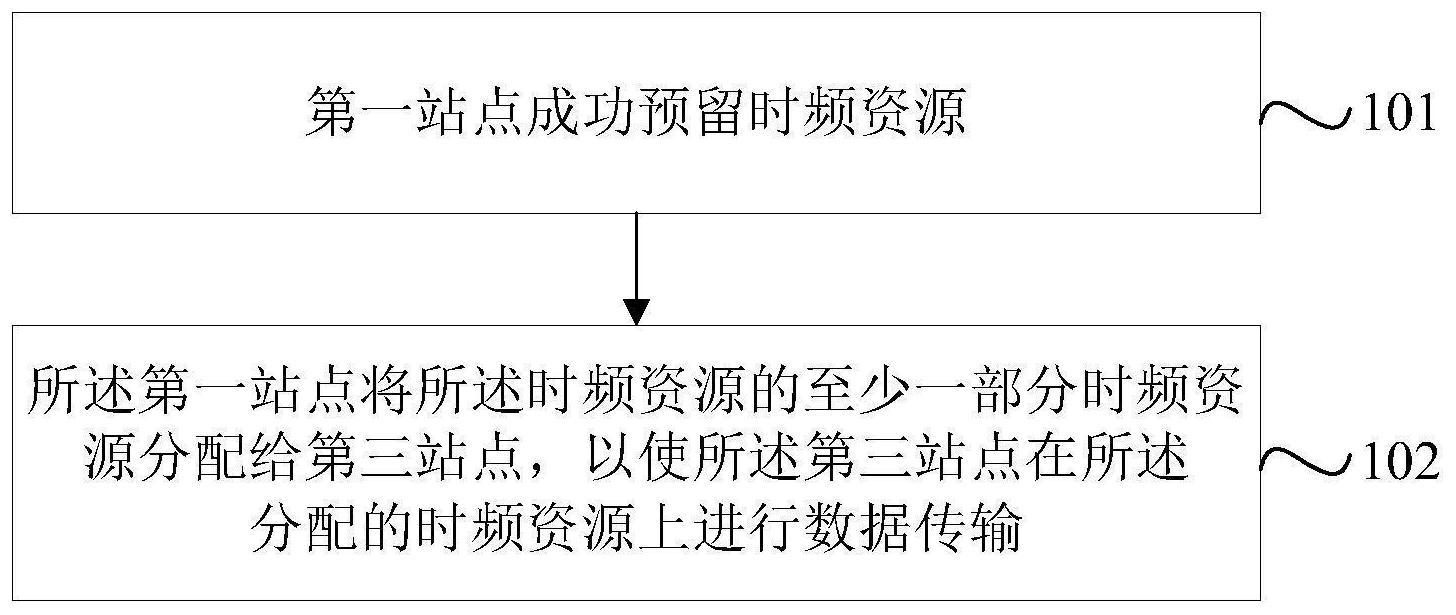
发明目的:本发明目的在于针对现有技术的不足,提供一种基于Raid-sharding的 GRA扩展算法,根据已经实现的复制方案以及特定对象的读写请求的变化,快速调整副本分 布以反映新的需求。 技术方案:本发明所述基于Radi-sharding架构数据副本的方法,包括如下步骤: (1)初始化种群:采用SRA算法初始化一半种群,随机初始化另一半种群,并混合两 种初始化种群; (2)交叉:利用两点交叉机制配对混合后的种群的染色体,在染色体配对后,随机 选择两个比特,并且交换它们之间的比特串或者交换染色体中除了它们之外的比特串; (3)变异:以概率μm翻转每一位来执行变异,μm取值为0.01~0.02; (4)选择:通过适应度值f对染色体进行质量评估,使用轮盘选择算法,将2πfi/f的 扇形区域分配给第i个染色体,创建后代,后代循环执行步骤(2)~(4)。 本发明进一步优选地技术方案为,步骤(1)初始化种群的具体步骤为: a)使用SRA算法初始化一半种群; b)由SRA产生的一半染色体收到其值得1/4的随机扰动; c)在所有随机决策中检查并维持染色体有效性; d)随机初始化另一半种群; 3 CN 111553458 A 说 明 书 2/3 页 e)混合两种初始化种群。 作为优选地,步骤(2)中混合后的种群的染色体在交叉时,随机确定并列部分,交 叉率为μc,μc取值为0.6-0.9。 优选地,在交叉完成后,将未交换的比特串进行交换。 优选地,步骤(3)中在执行变异时,对于会导致违反存储约束,以及会导致从主站 点取消分配对象的特定位跳过突变。 优选地,步骤(4)的具体步骤为: a)使用轮盘选择算法实现后代分配,将2πfi/f的扇形区域分配给第i个染色体; b)若生成数字在0到2π之间,则创建后代; c)根据简单选择算法的比例,适应度值得整数部分为染色体分配后代; d)从初始种群μ=Np开始,创建另两个同等大小的子群种,一个来自交叉,另一个 来自变异算子,三个种群的所有染色体竞争下一代的适应者; e)利用一代中最好的染色体取代最差的染色体,并允许精英染色体每五代复制一 次; f)将小数部分放入轮盘中,用于定义剩余的后代。 有益效果:本发明中使用Raid-sharding架构数据副本是为了定义适当的副本分 发,以最小化网络流量,将问题表述为约束优化问题,并表明相关决策问题是NP完全的;本 发明的算法给出了已经实现的复制方案以及特定对象的读写请求的变化,它可以快速调整 副本分布以反映新的需求,AGRA运行的搜索空间缩小,使其能够在动态环境时间限制内提 供可接受的高解决方案质量;本发明解决了数据复制问题,并制定了适用于的成本模型大 型分布式系统。 附图说明 图1为本发明的算法流程图。