技术摘要:
本发明属于底栖动物鉴定技术领域,公开了一种利用环境沉积物样品监测淡水底栖动物的方法,包括以下步骤:1)首先采集环境沉积物样本,清洗后保存;2)向步骤1)保存的环境沉积物样本中加入无水乙醇进行浸提处理,处理一段时间后取混合均匀的浸提液,将所述浸出液进行真空 全部
背景技术:
淡水大型底栖动物是流域生态健康评价的重点监测对象之一,目前的监测流程主 要为采集沉积物样本、人工挑拣、形态学鉴定,这一流程有着耗时长、人力成本高、需要专家 具有较高的鉴定水准等局限性。通过提取沉积物样品中的DNA,通过PCR扩增的方式对沉积 物的种类进行溯源的方式可以有效克服现有技术中利用形态学鉴定存在的耗时长,人力成 本高,操作繁琐的缺陷。 经检索,针对沉积物样品中生物的DNA提取方式,现有技术已有相关的申请案公 开,如中国专利申请号为200510079754.1,授权公告日期为2008年12月3日的申请案公开了 一种从海洋沉积物中同时提取微生物宏基因组DNA和总RNA的方法,包括将海洋沉积物用变 性溶液处理,加入沙子振荡,加入蛋白酶和溶菌酶进行裂解,再加入SDS和PVPP溶液进行进 一步裂解,加入萃取剂进行处理,萃取液用异丙淀,最后用核酸吸附树脂进行纯化。 再如中国专利申请号为CN200610136833.6,公开日期为2009-06-10的申请案公开 了深海沉积物总基因组的抽提方法,包括样品的处理和DNA的抽提。用无菌人工海水基本盐 溶解离心收集的深海沉积样品,向实验样品中加入高浓度细胞裂解液,在沸水浴中裂解细 胞,加入酚/氯仿/异戊醇混和液离心,重复抽提,收集上清液;上清液用无水乙醇沉淀,离心 收集沉淀;用乙醇洗涤沉淀物,沉淀物经真空干燥,溶解沉淀,即为基因组DNA提取液。该申 请案的方法运用微生物学、生物化学、分子生物学、物理学和化学的原理,以解决针对生境 特殊的深海微生物的总基因组DNA提取过程中存在的效率低、通用性差、产率低、花费高、样 品耗量大等问题,从而提高深海样品中DNA的产率和种类数量,使其分子生态学研究结果更 全面,更准确。 COI基因是动物细胞线粒体内细胞色素氧化酶亚基一的编码基因,其序列在物种 之间有较大差异,是常用的物种鉴定条形码区域之一。经检索,中国专利申请号 201910840869.X,公开日期为2020.03.20申请案公开了一种底栖动物COI基因的高通量测 序方法及其应用,该申请案的方法主要包括以下步骤:S1、将底栖动物混合样品的DNA样本 为模板,PCR扩增COI基因;S2、以S1步骤中的PCR扩增产物为模板,进行PCR扩增,扩增的引物 对包括正向引物和反向引物,所述引物对上包括焦磷酸测序高通量测序接头,每个样品对 应一种焦磷酸测序高通量测序接头,得到PCR产物作为DNA文库;S3、将S2步骤中获得的DNA 文库进行质量检测,然后选择合格的DNA文库进行emPCR扩增和高通量测序。该申请案的方 法测序方法通量高,一次性测序即可获得COI标准条形码基因~658bp的数据,准确率高,提 供了更大分类学分辨率。针对底栖动物样品,其实施例中公开的采集及处理方法为:首先, 利用索伯网或彼得森采泥器在不同生境采集底栖动物样品,每个点采集3个平行样本,清洗 粗挑后放入300mL标准采样品瓶内,95%酒精固定保存。使用组织基因组DNA提取试剂盒对 3 CN 111593099 A 说 明 书 2/7 页 上述准备的生物混合样品进行DNA提取,测定DNA浓度并统一稀释成终浓度5ng/μl。 基于现有技术的缺陷,亟需发明一种能够提高监测准确度,覆盖范围广的针对环 境沉积物样品中淡水底栖动物监测方法。
技术实现要素:
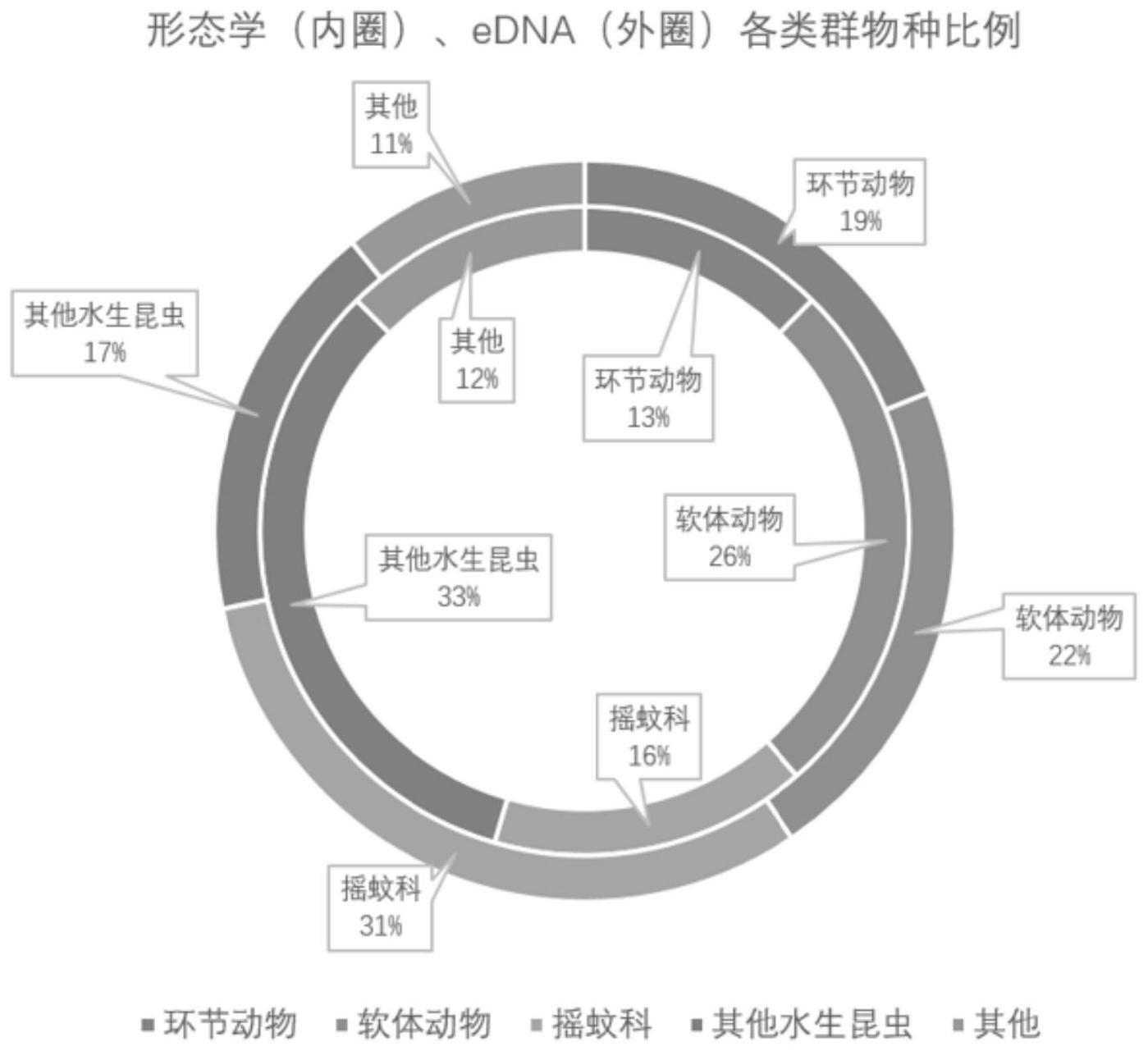
1.要解决的问题 现有技术中对环境沉积物样品中淡水底栖动物监测方法存在一次性采集量小,底 栖动物的覆盖度低,导致的监测结果准确度低的缺陷,本发明的方法将环境沉积物样品首 先采用无水乙醇浸提一段时间,使底栖动物DNA均匀释放到无水乙醇中,达到均质的状态, 从而将沉积物的固体样品转化成为均一化的溶液形式,通过采集含有DNA样品的溶液进行 DNA提取,可实现大范围环境样本的准确监测。 2.技术方案 为了解决上述问题,本发明所采用的技术方案如下: 本发明提供了一种利用环境沉积物样品中淡水底栖动物监测方法,包括以下步 骤: 1)首先采集环境沉积物样本,清洗后保存; 2)向步骤1)保存的环境沉积物样本中加入无水乙醇进行浸提处理,处理一段时间 后取混合均匀的浸提液,将所述浸出液进行真空离心,弃上清液,获得干燥的组织残渣,进 行DNA提取; 3)利用提取得到的DNA条形码片段进行扩增,得到扩增产物; 4)将步骤3)得到的扩增产物进行测序与分析,得到底栖动物的物种信息。 作为本发明更进一步的改进,所述步骤1)将清洗后的样本在-20℃~-80℃冷冻保 存,冷冻时间为1-3天。 作为本发明更进一步的改进,所述步骤2)中浸提的时间为3~30天。 作为本发明更进一步的改进,所述步骤2)中,按照1g沉积物换算为1ml的换算方式 向沉积物样本中加入3~5倍体积的无水乙醇。 作为本发明更进一步的改进,所述真空离心条件为:在300~1000rpm,37℃条件下 离心2~3小时。 作为本发明更进一步的改进,步骤2)中使用组织DNA提取试剂盒对所述组织残渣 进行DNA提取,再测定DNA浓度。 作为本发明更进一步的改进,步骤2)中使用OMEGA Tissue试剂盒对所述组织残渣 进行DNA提取;使用Qubit2.0或NANODROP 2000平台测定DNA浓度。 用于扩增和测序的DNA条形码为COI基因上一段长约313bp的片段,该片段被广泛 运用于大型底栖动物的DNA条形码研究。 其上游引物序列为:mlCOIintF:GGWACWGGWTGAACWGTWTAYCCYCC; 其下游引物序列为:jgHCO2198:TAIACYTCIGGRTGICCRAARAAYCA。 作为本发明更进一步的改进,使用mlCOIintF-dgHCO2198引物对进行PCR扩增,所 述PCR扩增条件为: S1、构建PCR扩增体系:25μl PrimeStarMax扩增酶,5μl DNA模板,所述DNA模板总 4 CN 111593099 A 说 明 书 3/7 页 量约20ng,上下游引物各1.25μl,无酶水17.5μl; S2、98℃预变性3分钟; S3、98℃变性15秒,46~48℃复性30秒,72℃延伸45秒,共重复35个循环; S4、72℃延伸5分钟,得到PCR扩增产物。 作为本发明更进一步的改进,所述步骤4)包括以下步骤: 4-1)、通过高通量测序技术对所述扩增产物测序; 4-2)、测序结果在qiime、usearch等软件平台上处理,对照NCBI数据库进行物种注 释; 4-3)、将所述物种注释结果进行人工筛检,作为生物监测结果。 得到的PCR扩增产物用浓度为2%的琼脂糖凝胶电泳检测。 作为本发明更进一步的改进,根据步骤4)的生物监测结果进一步整理得到底栖物 种名单。 3.有益效果 相比于现有技术,本发明的有益效果为: (1)本发明的利用环境沉积物样品监测淡水底栖动物的方法,首先将沉积物样品 采用无水乙醇浸提一段时间,能够使样品中底栖动物的DNA均匀释放到无水乙醇中,达到均 质的状态,从而将原本为固体形态的沉积物样本转化成为溶液形式,再对溶液形式的DNA样 本进行离心浓缩,进而DNA提取,这种前处理的方式更大程度上保证了样品的均匀性,可以 通过采集少量的样品实现大范围的底栖动物监测,而现有技术中直接对采集得到的固体样 品进行离心处理的方式,需要采集的大量的生物样品,且容易导致不均匀性。因此本发明的 方法有效克服了现有技术在环境样品采集过程中存在的一次性采集量小,底栖动物的覆盖 度低,导致的监测结果准确度低的缺陷。 (2)本发明的利用环境沉积物样品监测淡水底栖动物的方法,首先对沉积物样本 进行冷冻处理,再解冻处理,可以使经过冷冻处理的底栖动物细胞和组织更迅速的脱落,进 而使更多样本释放到无水乙醇浸提液中,保证提取的DNA样本浓度,从而更好的实现扩增需 求。 (3)本发明的利用环境沉积物样品监测淡水底栖动物的方法,使底栖动物的鉴定 速度大大加快。一次100个样点的流域监测,样品前处理和鉴定流程,按照现有的方法大约 需要约4人的小组35个工作日,而利用本发明的监测方法,仅需1-2人约7个工作日即可完成 监测过程。 (4)本发明的利用环境沉积物样品监测淡水底栖动物的方法,监测结果可靠性强。 现有技术中采用形态学监测,难以保证结果的统一性:不同专家、不同操作条件,可能对同 一物种产生不同的鉴定结果,本方法采取标准化的分子生物学和生物信息学流程,不同样 品、不同人员的实验结果具有统一性,有利于大范围时间序列和空间序列的整体研究。 (5)本发明的利用环境沉积物样品监测淡水底栖动物的方法,提供的监测流程,包 含环境样品采集、样品保存、样品前处理、分子生物学实验、生物信息学分析等一系列步骤, 为淡水大型底栖动物的快速监测提供了完整且易于操作的指导,可用于环境例行监测、科 学研究等多个场景。 (6)本发明的利用环境沉积物样品监测淡水底栖动物的方法,利用无水乙醇浸提 5 CN 111593099 A 说 明 书 4/7 页 的前处理方式针对底栖动物监测具有更好的针对性,对提取的样品采用mlCOIintF- dgHCO2198引物对进行PCR扩增,得到的PCR扩增结果表明大多数样品呈现出明亮、清晰的条 带,实现了目标物的扩增,而且物种覆盖度高,能够监测获得形态学上极难监测实现的13种 属于仙女虫科的物种,更进一步说明了本发明方法监测范围的广度。 (7)本发明的利用环境沉积物样品监测淡水底栖动物的方法,通过对比了形态学 检测出各物种生物量与本方法检测出各物种序列数的定量关系,发现多个物种的生物量相 对丰度对数值与序列数相对丰度对数值存在显著正相关关系(p<0.01),说明了本发明方法 的可行性与可靠性。 附图说明 图1为实施例1中对COI基因文库的检测的凝胶图谱,其中图1A~图1D分别展示了 不同采集位点提取得到的DNA凝胶图谱; 图2为实施例1中各类群序列注释结果占比; 图3为实施例1中各类群OUT数目占比; 图4为实施例1中形态学检测与eDNA检测各类群物种比例对比; 图5为实施例1中形态学检测与eDNA检测定量关系拟合结果对比;图5A分别展示了 颤蚯科和摇蚊科的生物量相对丰度对数值与序列数相对丰度对数值存在显著正相关关系, 图5B展示了环棱螺属的生物量相对丰度对数值与序列数相对丰度对数值存在显著正相关 关系; 图6为对比例A、对比例B和实施例1的三种方法得到的DNA浓度比较; 图7为实施例1的方法与对比例A中两种方法所得序列来源的比较。