技术摘要:
本发明属于生物领域,尤其设计一种高通量的病原体微生物基因检测筛查方法。一种高通量的病原体微生物基因检测筛查方法,包括:S1:设计探针;S2:芯片合成;S3:提取样品中的DNA或RNA;S4:文库构建;S5:文库目标区域杂交捕获和测序;S6:采用高通量测序平台检测;S7 全部
背景技术:
病原体微生物的检测和鉴定对临床上判断感染类型及进行有针对性的治疗都具 有非常重要的意义。病原体微生物种类繁多,来源广泛,而即使是不同种类的病原体感染, 表现在临床症状上具有一定的相似性,比如普遍伴有发烧等症状,所以很难从临床表现区 分感染类型,而准确有效的筛查或诊断检测方法具显得至关重要。目前临床上采用的病原 体微生物检测方法主要有培养法,基于蛋白的抗原抗体特异性检测,以及基于核酸的基因 检测,常见的有QPCR,一代测序和高通量测序等等。由于培养条件的局限性,可被培养的微 生物种类较少,而且培养需要较长的时间周期,培养法的劣势比较明显。基于抗原抗体特异 性检测具有速度快的优势,但能够检测的微生物种类不多,而且无法准确地确定具体的病 原体微生物的物种。核酸检测具有较强的普遍性,在病原体微生物的检测上有着越来越广 的应用。 病原体微生物的基因检测方法中,QPCR是应用最多的一种技术,通过选取微生物 基因组中物种特异的序列,设计相应的引物或探针,通过相对定量的方法来确定样品中是 否存在该病原体的感染,并计算病原体的基因组组拷贝数。QPCR对检测已知的病原体,有较 大优势,成本低,速度快,准确率高。但临床上,多种病原体可能会有类似的感染症状,而 QPCR的检测位点有限,需要多次检测才能满足对多种病原体实现检测的需求。同时,还有一 些罕见或未知的病原体,又会引发临床症状,这些感染类型QPCR很难解决。 由于高通量测序成本的下降,以及目标区域捕获技术的发展,基于高通量测序的 病原体感染检测也被越来越多地用到。目前常见的检测方式主要分为全基因组或宏基因组 (WGS)和目标区域捕获测序两类。宏基因组测序不作任何假设前提,对待测样品中的所有物 种的基因组都进行检测,虽然检测的范围很广,但是要求的数据量很大,并且其中大部分的 数据都是来自背景的序列,数据有效率很低,检测的灵敏度有限,成本又非常高。 目标区域捕获测序,事先将一系列病原体的物种特异序列设计成探针或引物,对 待测样品中的DNA进行目标区域富集,再进行测序分析。目标区域捕获测序常用的方法主要 有液相杂交捕获,和多重PCR两类。目标区域捕获测序对目标区域进行了富集,检测的灵敏 度相比全基因组测序大大提高,同时需要的测序数据量也较小,成本较低。但它只针对事先 确定的病原体微生物进行检测,检测范围存在一定的局限性。
技术实现要素:
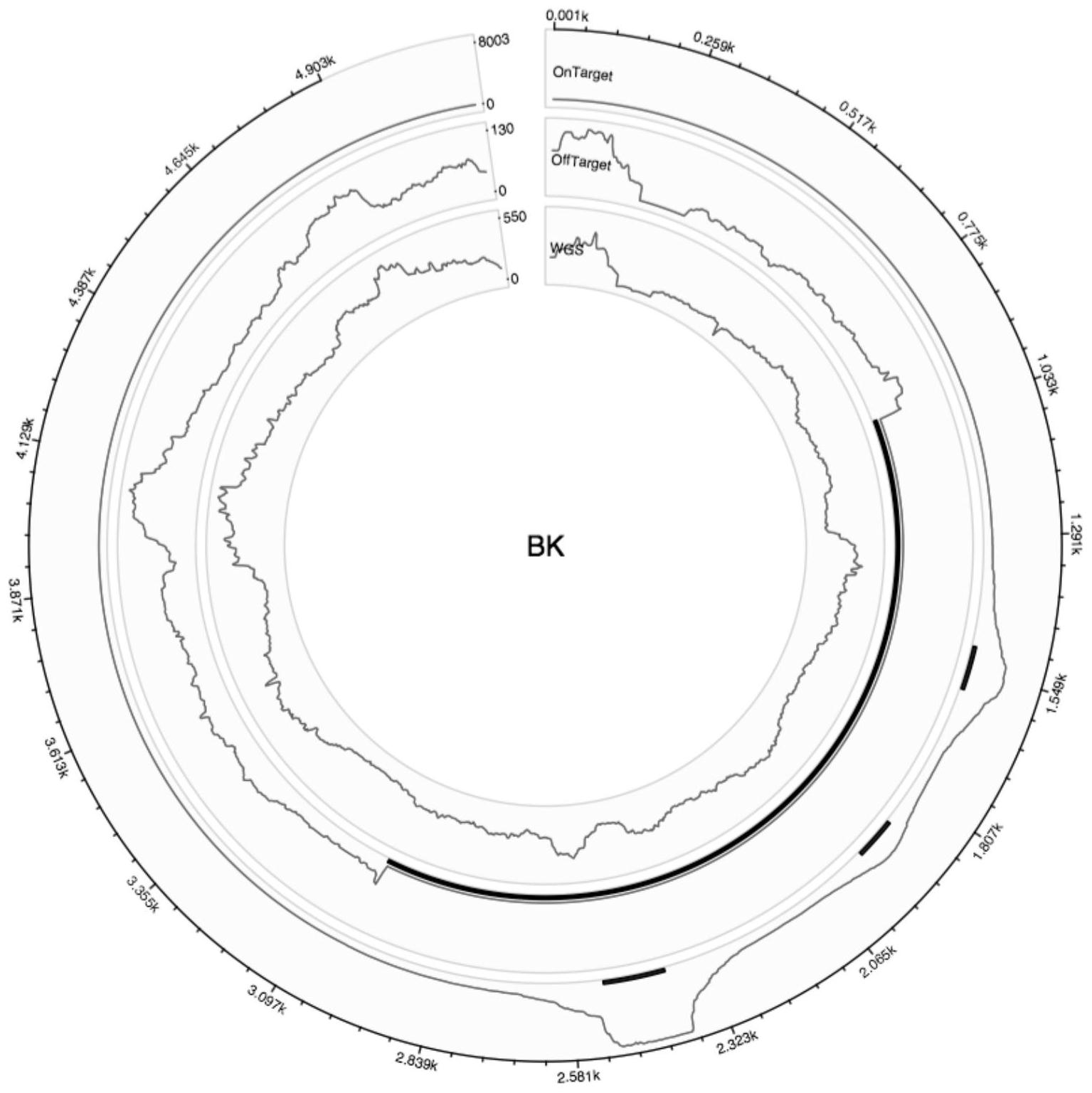
本发明的目的是通过液相杂交技术和高通量测序技术,同时实现对已知和未知病 原体的筛查。既可以对目标的病原体进行捕获和富集,提高检测的灵敏度,又可以通过对 “非目标区域”的序列进行分析,扩大检测范围。 具体的,本发明的技术方案如下: 3 CN 111549109 A 说 明 书 2/9 页 本发明第一个方面公开了一种高通量的病原体微生物基因检测筛查方法,包括: S1:设计探针; S2:芯片合成; S3:提取样品中的DNA或RNA; S4:文库构建; S5:文库目标区域杂交捕获和测序; S6:采用高通量测序平台检测; S7:进行生信分析,采用Samtools来计算病原体基因组序列每个位点的深度;与探 针目标区域重叠的序列被视为“目标序列”;相反,则被视为“非目标序列”。 应该理解,本发明不限于上述步骤,还可以包含其他的步骤,例如在步骤S1之前、 步骤S1和S2之间、步骤S2和S3之间、步骤S3和S4之间、步骤S4和S5之间、步骤S5和S6之间、步 骤S6和S7之间和步骤S7之后,还包含其他额外的步骤,而不超出本发明的保护范围。 优选的,选择目标病原体微生物作为筛查对象,运用生物信息学方法,选择该病原 体基因组上的物种特异序列,并以此区域设计探针序列。 优选的,每种微生物设计1-100条探针,探针合成后,等摩尔体积混合,得到液相检 测芯片。 优选的,所述样品为血液样品,样品采集和分离后得到cfDNA。 优选的,所述S4包括: S41:分别取cfDNA进行QuantiFluorTM-ST(Promega)定量和Agilent 2100检测质 量; S42:以cfDNA作为样本,使用二代构建文库测序试剂盒分别制备基因组DNA文库和 cfDNA文库。 更优选的,所述S42依次包括以下步骤: 末端补平、补平后纯化、加A尾、加A尾后纯化、加单分子标记接头、加接头后磁珠纯 化、文库扩增、文库鉴定和文库纯化步骤。 优选的,所述高通量测序平台为illumine X-Ten。 优选的,所述S7包括: S71:对下机数据进行数据分析,先进行数据拆分,再对数据进行质量值过滤,去除 低质量数据; S72:将测得序列的K-mers比对到所有参考基因组,将Kraken用作分类学分类器; 根据kraken的算法,建立自定义分类,得到数据库; S73:采用Samtools来计算病原体基因组序列每个位点的深度;得到目标序和“非 目标序列”; S74:使用自定义脚本来计算探针目标区域的覆盖范围以及非目标区域的序列数; 如果一个病原体的序列同时出现在目标区域和非目标区域,则认定为阳性。 优选的,所述病原体为BK病毒。应当理解,本发明检测的病原体并不限于BK病毒, 任何可用本发明方法检测出的病原体均在本发明的保护范围之内。 本发明第二个方面公开了上述方法在病原体检测中的应用。 在符合本领域常识的基础上,上述各优选条件,可任意组合,而不超出本发明的构 4 CN 111549109 A 说 明 书 3/9 页 思与保护范围。 本发明的技术方案使用液相杂交捕获技术,可以对要检测的目标病原体基因组序 列进行探针的设计和制作,并对检测样品进行目标区域的捕获测序。本发明技术方案,可以 通过调整杂交捕获的实验条件(杂交温度和杂交捕获试剂的配比),来获得不同比例的“上 靶率”。上靶率=(属于目标区域的序列总数)/(属于该样品的所有下机序列)。上靶率一般 是用来衡量液相杂交捕获技术的富集效率,上靶率越高,测序结果中属于目标区域的序列 比例就越高,富集效果越好。非目标区域的片段,往往是被当成“没有用的序列”而丢弃。而 本方面技术方案,可以对液相杂交捕获的测序结果中,属于“目标区域的序列”和“非目标区 域的序列”都能进行分析利用。通过调整杂交捕获条件,获得的“非目标区域的序列”非常接 近一个随机的WGS文库,而“目标区域序列”则是我们想要得到的目标片段。我们通过控制好 液相杂交捕获技术的“上靶率”,让测序结果中的“目标序列”和“非目标序列”合理分配。在 测序结果中,“目标序列”承担目标病原体的富集功能,能够以100倍以上的效率富集病原体 序列,提高检出灵敏度。结果中的“非目标序列”是几乎没有偏向的宏基因组数据(WGS),即 使没有被设计探针的病原体序列,只要存在于检测样品中,也能从“非目标序列”中被检测。 而目标病原体的序列,既能在“目标序列”中被富集,又能在“非目标序列”中检测上,从而提 高检测的准确率。 本发明相对于现有技术具有如下的显著优点及效果: 本发明方法既可以对目标的病原体进行捕获和富集,提高检测的灵敏度,又可以 通过对“非目标区域的序列”进行分析,扩大检测范围。通过调整液相杂交捕获流程,可以对 捕获过程的“on-target rate(上靶率)”进行调整,从20-75%不等。例如当上靶率在60% 时,会有60%的下机数据属于我们设计的目标病原体,而剩余的40%下机数据是对检测样 品核酸序列的随机测序,相当于一个宏基因组数据,而其中包含了非靶向的病原体序列,这 是对目标区域捕获测序的补充。 因此,通过一次检测,可同时实现“靶向病原体检测”和“宏基因组检测”,既能提高 靶向病原体的检测灵敏度,又能检测到未设计探针的病原体,扩大检测范围。 附图说明 图1为本发明实施例中检测一例BK病毒阳性的病人血浆样品的结果对比图。