技术摘要:
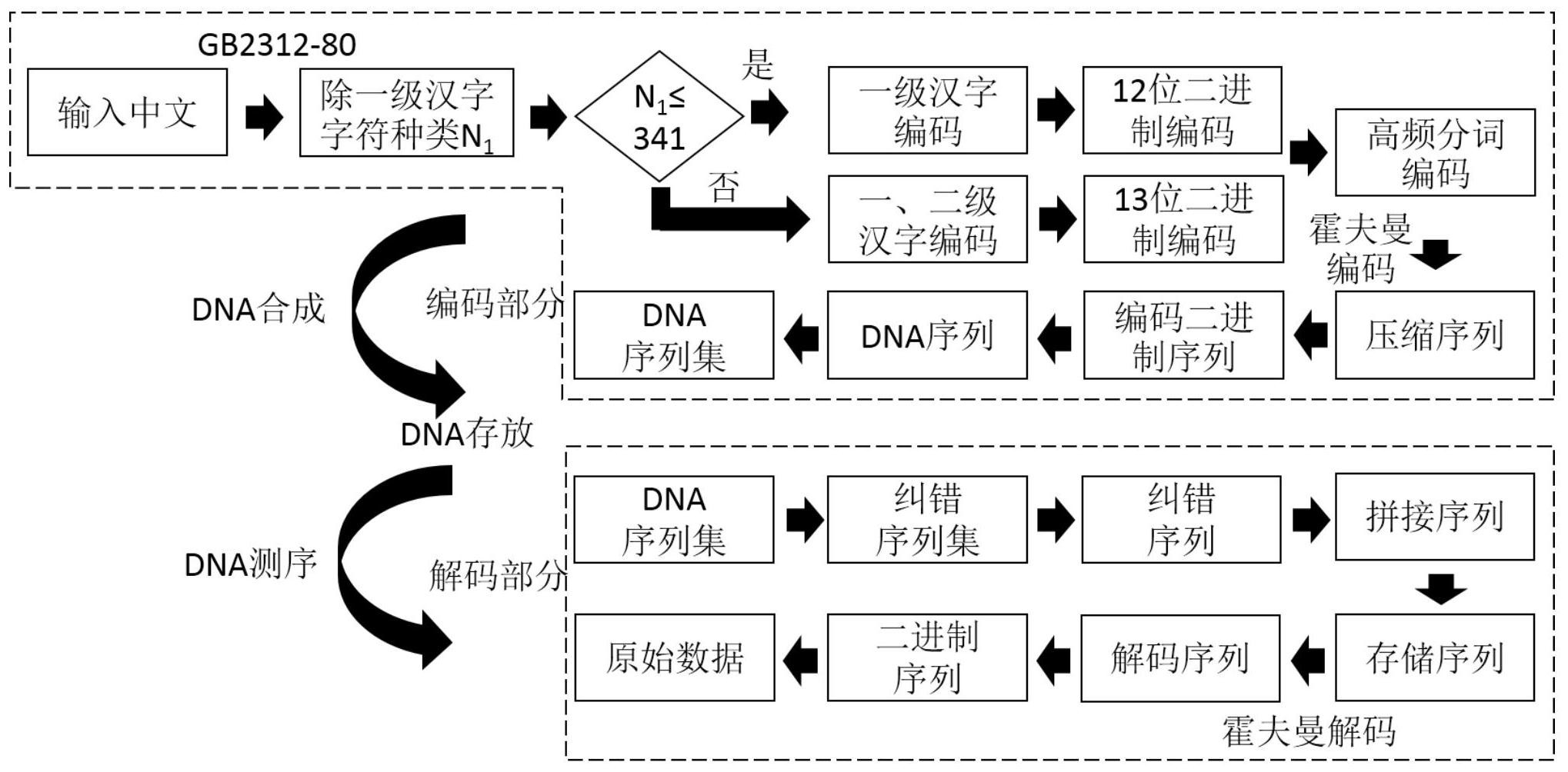
本发明公开了一种优化中文存储的DNA存储编码方法,包括如下步骤:1)输入中文文本,根据包含字符种类和GB2312‑80标准,对一级汉字或一、二级汉字重新编码。2)统计文本中分词出现频率,将出现频率乘以分词长度,并对乘积进行排序,对排在前列分词进行编码。3)所有字 全部
背景技术:
全球数据信息总量已达到30ZB,并将很快超过现有硬盘等存储介质的承受能力。 DNA数据存储技术开辟了一种新的存储模式,其发展对于节省存储能源及推进大数据存储 发展有着重要作用。DNA数据存储近年来逐渐成为全球研究的热点。包括哈佛大学、哥伦比 亚大学、微软研究院、华盛顿大学和剑桥大学等国内外多家研究机构均展开对DNA存储的研 究。 单位质量的DNA约有1021个碱基,可存储455EB信息,此信息量为全球一年信息总 量的1/4;单位体积的DNA可存储的信息为整个互联网的33倍。DNA单位体积的存储密度是硬 盘和存储器的106倍,是闪存的103倍。DNA作为最稳定的储存设备之一,对于外部环境,如高 温、震荡等具有极强的抗干扰能力。 DNA存储编码模型的研究面临的困难是存储效率与合成难度、成本之间的矛盾。存 储效率越高,合成难度越大,成本越高,甚至难以合成。目前相关研究均处于初级阶段,数据 量较小,且主要是对英文文本、视频、图像等进行编码存储,对中文的编码存储较少。传统的 中文编码一个汉字占据2个字节,转换为碱基序列为8个碱基,冗余度大。传统编码算法对中 文文本的适应性差,编码压缩率较低。以DNA存储领域使用最为广泛的霍夫曼编码为例,该 算法对英文文本效果较好,但对中文文本效果很不稳定,总体较差。 针对以上问题,进行基于中文的优化编码,并融合数据压缩算法和纠错编码,降低 了中文文本的冗余度,提高了DNA存储编码压缩效果,获得了极高的中文编码潜力。
技术实现要素:
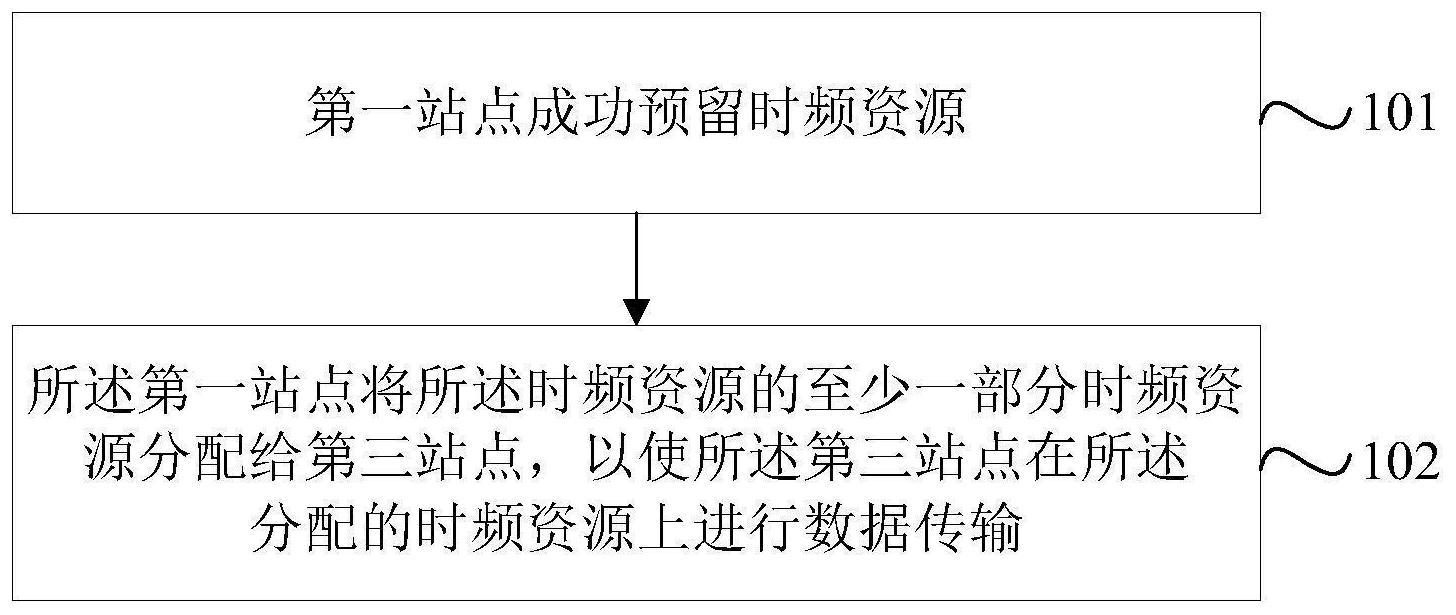
技术问题: 针对现有DNA存储模型对于中文的存储效率不高,冗余度大的问题,通过一种中文 优化编码方案,降低中文文本的冗余度,提高DNA存储编码压缩效果。 技术方案: 本专利提供一种优化中文存储的DNA存储编码方法,包括如下步骤。 编码过程: 1)根据GB2312-80《信息交换用汉字编码字符集》,将其中的一级汉字按顺序重新 编号为0至3754。 2)输入待编码中文文本,根据文本包含的字符种类不同,设计两种字符编号方式: ①编号方式E1:统计其中出现的一级汉字以外的字符种类数,若不超过341种,文 本中所有的一级汉字以外的N1种字符编号为3755至3755 N1-1,N1≤341,进入步骤3)。 ②编号方式E2:若一级汉字以外的字符种类数超过341种,将GB2312-80中的二级 汉字按顺序重新编号为3755至6762,文本中所有的一级和二级汉字以外的N种字符编号为 3 CN 111600609 A 说 明 书 2/5 页 6763至6763 N2-1,N2≤1429,进入步骤3)。 3)统计文本中分词的出现频率,每个分词包含的字符数目为2到4个。将每个分词 的出现频率乘以分词的长度(包含的字符数目),并将乘积进行降序排列。 ①采用编号方式E1,选择乘积值最大的前341-N1(N1≤341)个分词,依次编号为 3755 N1到4095; ②采用编号方式E2,选择乘积值最大的前1429-N2(N2≤1429)个分词,依次编号为 6763 N2到8191。 4)将文本中的所有字符转换为对应的数字编号,然后将数字转换为二进制序列, 二进制数的位数根据步骤2)的编号方式确定。 ①采用编号方式E1,每个数字转换为12位二进制数。 ②采用编号方式E2,每个数字转换为13位二进制数。 5)对二进制序列进行霍夫曼编码压缩,然后选择一种DNA四进制模型,将二进制序 列转换为DNA序列,并划分为等长若干列,每列列首添加地址码。每列包含100个碱基,列首 添加9位地址码(1个碱基的文件码和8个碱基的编号码)。 6)采用RS编码对每一列RS编码纠错,在列尾添加纠错码(18个碱基),得到包含127 碱基的DNA序列。 7)将得到的所有序列按文件码和编号码排序,每123行组成一个123行*127列的矩 阵,其中123行表示123条已完成编码的连续编号DNA序列,127列表示DNA序列的127个碱基。 然后,从存储位置(第10列)开始,逐行进行RS编码纠错,纠错码长度为18个碱基,生成127* 127的矩阵,即每123行DNA序列后,添加4行纠错序列。纠错序列前9列添加独立索引,按步骤 5)选择的DNA存储四进制模型编码。 解码过程: 1)解码过程为编码的反向过程,将测序得到的DNA序列按文件码和编号码排序,纠 错序列按顺序插入其中,重新构建127*127矩阵,首先进行RS编码的行解码,根据4行纠错序 列逐行对错误碱基进行纠正;然后对123行DNA序列,每一行进行RS解码。 2)按照地址码拼接,然后删除地址码和纠错码。 3)根据选择的四进制模型,将DNA序列转换为二进制序列。 4)对二进制序列进行霍夫曼解码,得到初始二进制序列。 5)根据选择的编码方式E1或E2,将二进制序列按12或13位划分,并重新生成输入 文件。 有益效果:本发明方法引入中文优化编码,并结合压缩算法(霍夫曼编码)和RS纠 错码,成功完成DNA存储中文数据的编码、合成、存储、测序、解码的完整流程,提高了中文文 本的编码潜力。 1 .相较于现有的基于英文文本的传统压缩算法,有效降低了中文文本的数据冗 余。 2.引入压缩与纠错算法,在提高数据存储效率的同时,对数据存储和读取过程中 的错误进行有效纠正。 4 CN 111600609 A 说 明 书 3/5 页 附图说明 图1为本发明的方法流程示意图; 图2为DNA序列组成及RS码纠错示意图; 图3为实施例一的输入文本示例; 图4为实施例二的输入文本示例;