技术摘要:
本发明提供了一种厚壳贻贝不同发育阶段的荧光定量内参基因及其引物和应用,厚壳贻贝不同发育阶段的荧光定量内参基因为RPS23、CYTB5、EF‑1α和α‑Tubulin,RPS23基因的碱基序列如SEQ ID NO.1所示,CYTB5基因的碱基序列如SEQ ID NO.2所示,EF‑1α基因的碱基序列如SEQ I 全部
背景技术:
厚壳贻贝(Mytilus coruscus)隶属软体动物门双壳纲贻贝目,是一种近海暖温性 的底栖贝类,是我国继牡蛎、蛤和扇贝后的第四大海产贝类。贝类养殖不仅满足消费者对于 鲜活、优质海产品的需求,同时对国家蓝色海洋可持续发展起着非常重要的作用。厚壳贻贝 作为附着性贝类,幼体时期需要经历一个附着和变态的过程,也是其从浮游幼虫的生活方 式转变为底栖生活方式的重要转变过程。幼体的变态过程主要由外界环境因子和内源性因 子所控制,关于外界环境因子如海洋微生物被膜、海洋细菌被膜和人工诱导物等已有大量 的研究报道。近年来,在分子水平阐明幼虫变态发育的调控机制受到科学家们的广泛关注。 实时荧光定量PCR(quantitative real-time polymerase chain reaction,qRT-PCR)是一 种定量检测目的基因表达的技术,可以捕捉到PCR扩增反应中每个循环产物产生的荧光信 号,完成定性以及定量分析。与传统的PCR技术相比,它具有高精确度、强特异性、快速检测 和重复性好等优点。 内参基因是指在对某一个目的基因进行定量研究时,在不同实验条件下都能在待 测样品中实现稳定表达的一类基因,其作用是校正目的基因的定量过程。这些内参基因一 般都是管家基因,在细胞内可以稳定表达,从而有助于细胞各种功能的实现。在进行qRT- PCR过程中,待测样品可能会在反转录效率上存在差异,所以需要选择可以稳定表达的内参 基因作为校正标准。 公开号为CN104073557A的专利中,筛选出的β-actin、GAPDH和RS18是Poly(I:C)应 激实验中,牡蛎血细胞内表达最稳定的3个内参基因,因此可以同时使用做为荧光定量内参 基因。此外,一些内参基因只在特定时期或处理条件下稳定表达。发育时期或处理条件不 同,常用内参基因的表达就会产生巨大差异。因此,对于不同的实验条件和样本类型,需要 不同的内参基因。
技术实现要素:
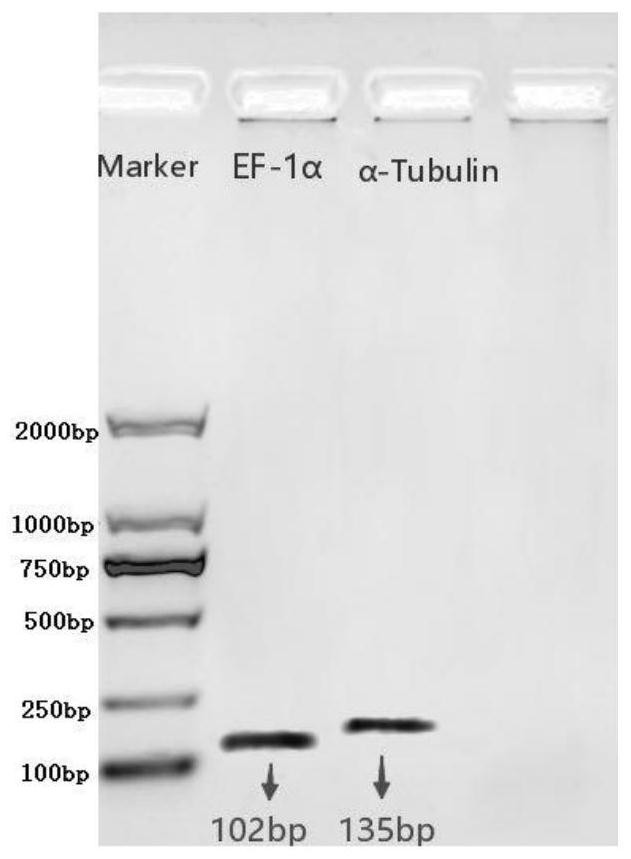
针对现有技术中的不足,本发明的首要目的是提供一种厚壳贻贝不同发育阶段的 荧光定量内参基因,即筛选厚壳贻贝(Mytilus coruscus)不同生长发育阶段的荧光定量内 参基因。基于荧光定量PCR中的绝对定量法在厚壳贻贝不同生长发育阶段相关实验中对其 他功能基因进行定量分析所需的内参基因及其引物序列,从而筛选出稳定表达的内参基 因,进一步大大提高qRT-PCR定量的准确性。 本发明的第二个目的是提供一种厚壳贻贝不同发育阶段的荧光定量内参基因的 引物。 本发明的第三个目的是提供一种厚壳贻贝不同发育阶段的荧光定量内参基因和 3 CN 111575390 A 说 明 书 2/9 页 引物的应用。 为达到上述目的,本发明的解决方案是: 厚壳贻贝不同发育阶段的荧光定量内参基因,荧光定量内参基因为RPS23、CYTB5、 EF-1α和α-Tubulin,RPS23基因的碱基序列如SEQ ID NO .1所示,CYTB5基因的碱基序列如 SEQ ID NO.2所示,EF-1α基因的碱基序列如SEQ ID NO.3所示,α-Tubulin基因的碱基序列 如SEQ ID NO.4所示。 为达到上述第二个目的,本发明的解决方案是: RPS23、CYTB5、EF-1α和α-Tubulin内参基因的特异性引物中RPS23基因的引物序列 为: RPS23-F:5’-TCACAGCCTTCGTCCCTAA-3’, RPS23-R:5’-CCTTTGAACAGAGCCCAGA-3’。 CYTB5基因的引物序列为: CYTB5-F:5’-CACTCAACTGACGCAAGAGAT-3’ CYTB5-R:5’-CAGCCAATGCCACACCAA-3’。 EF-1α基因的引物序列为: EF-1α-F:5’-CACCACGAGTCTCTCCCTGA-3’ EF-1α-R:5’-GCTGTCACCACAGACCATTCC-3’。 α-Tubulin基因的引物序列为: α-Tubulin-F:5’-TTGCAACCATCAAGACCAAG-3’ α-Tubulin-R:5’-TGCAGACGGCTCTCTGT-3’。 为达到上述第三个目的,本发明的解决方案是: 厚壳贻贝不同发育阶段的荧光定量内参基因在厚壳贻贝荧光定量中的应用。 RPS23基因的引物序列在厚壳贻贝荧光定量中的应用。 CYTB5基因的引物序列在厚壳贻贝荧光定量中的应用。 EF-1α基因的引物序列在厚壳贻贝荧光定量中的应用。 α-Tubulin基因的引物序列在厚壳贻贝荧光定量中的应用。 具体步骤为: 1.内参选择和引物设计: 根据本实验室厚壳贻贝转录组数据库,选择4个常用的管家基因作为候选内参基 因,分别是RPS23、CYTB5、EF-1α和α-Tubulin。首先利用引物设计软件Primer Premier 5.0 设计这4个候选内参基因的引物。 2.内参基因PCR扩增 4个候选内参基因RPS23、CYTB5、EF-1α和α-Tubulin以厚壳贻贝不同发育阶段的 cDNA为模板进行PCR扩增。经PCR扩增后,用1%的凝胶琼脂糖电泳检测其扩增产物的完整 性。若出现单一且明亮的条带,证明扩增产物完整。再使用Nanodrop 2000紫外分光光度计 对DNA浓度与纯度进行检测,测定A260/A280和A260/A230比值,最后送公司测序(生工生物 工程(上海)股份有限公司),测序结果与转录组基因序列完全匹配。 3.割胶回收及产物纯化 使用验证后的引物重新进行PCR扩增,产物进行1%的凝胶琼脂糖电泳,然后在凝 4 CN 111575390 A 说 明 书 3/9 页 胶成像仪下切割目的条带,进行割胶回收。用SanPrep柱式DNA胶回收试剂盒,将溶解的DNA 按说明书纯化,然后将获得的纯化PCR产物作为标准品。最后使用Nanodrop 2000紫外分光 光度计对DNA浓度与纯度进行检测,测定A260/A280和A260/A230比值,记录DNA浓度。 4.实时荧光定量PCR 利用RNAiso Plus试剂提取厚壳贻贝不同发育阶段的总RNA,根据试剂盒说明书去 除基因组DNA,并进行反转录。转录后产物稀释100倍并冻存于-20℃冰箱中,用作qRT-PCR模 板。将纯化后的标准品按照10倍比例稀释成7个梯度,获得7组不同浓度的cDNA,并将其作为 模板进行qRT-PCR检测,得到候选基因的标准曲线。之后再用qRT-PCR检测4个候选基因在厚 壳贻贝不同发育阶段中的表达。 5.数据分析 本实验利用geNorm、NormFinder、BestKeeper3个常用内参基因分析软件以及Ct值 分析法对qRT-PCR产生的原始Ct值进行数据分析。 由于采用上述方案,本发明的有益效果是: 本发明从4个常用候选内参基因中筛选得到2个在厚壳贻贝不同发育阶段中表达 相对最稳定的内参基因,以应用于厚壳贻贝不同发育阶段幼体的基因表达分析研究,选取 稳定的内参基因是荧光定量qRT-PCR实验中重要的一环,能显著提高qRT-PCR所得数据的准 确性,从而为厚壳贻贝不同发育阶段幼体实验中功能基因表达的精确定量提供有力保证。 附图说明 图1为本发明的RPS23的凝胶电泳结果示意图。 图2为本发明的CYTB5的凝胶电泳结果示意图。 图3为本发明的EF-1α和α-Tubulin的凝胶电泳结果示意图。 图4为本发明的geNorm法分析厚壳贻贝不同发育时期候选内参基因稳定性结果: (a)候选内参基因稳定值;(b)候选内参基因平均表达稳定性结果。 图5为本发明的NormFinder法分析厚壳贻贝不同发育时期候选内参基因稳定性结 果。 图6为本发明的厚壳贻贝不同发育时期中CYTB5的Ct值分布。 图7为本发明的厚壳贻贝不同发育时期中RPS23的Ct值分布。 图8为本发明的厚壳贻贝不同发育时期中α-Tubulin的Ct值分布。 图9为本发明的厚壳贻贝不同发育时期中EF-1α的Ct值分布。